50
n.11 settembre 2013
Tecniche biomolecolari
utilizzate ai fini
del biomonitoraggio
Sono state sviluppate e
ottimizzate diverse tecniche
biomolecolari che consentono
di identificare e quantificare
sia la presenza che l’attività
biodegradativa di microrganismi
decloranti coinvolti nella
declorazione riduttiva in matrici
ambientali contaminate. Le
metodologie adatte a questo
scopo (1- 5) prevedono l’utilizzo
combinato di tecniche basate
sull’ibridazione fluorescente
in situ (Fluorescence In Situ
Hybridization, Fish e CAtalyzed
Reporter Deposition Fish,
Card-Fish) e sull’analisi di geni
catabolici in microrganismi
coinvolti nei processi di
interesse (Real Time PCR,
qPCR e Reverse Transcription
qPCR, RT-qPCR).
Quantificazione mediante
ibridazione fluorescente
in situ: Fish e Card-Fish
Le tecniche di ibridazione
fluorescente in situ consentono
di quantificare i batteri
di interesse attraverso
l’utilizzo di sonde molecolari
oligonucleotidiche fluorescenti
che li rendono chiaramente
visibili in microscopia a
epifluorescenza. La differenza
tra la Fish e la Card-Fish
risiede soltanto nella tipologia
di fluoroforo legato alla sonda
e nella possibilità di rilevare
microrganismi con diverso
grado di attività metabolica:
con la Fish è possibile rilevare
microrganismi con un elevato
livello di attività metabolica
(l’intensità del segnale di
fluorescenza è elevata quando
i microrganismi sono attivi)
mentre con la Card-Fish si
rilevano anche microrganismi
caratterizzati da una bassa
attività metabolica (l’intensità
del segnale di fluorescenza è
sempre al massimo e consente
di visualizzare allo stesso modo
anche cellule con diverso grado
di attività metabolica). Il dato
quantitativo che si ottiene in
seguito all’applicazione delle
tecniche Fish e Card-Fish è
espresso come numero di
cellule (n°) per litro (L) di acqua
o grammo (g) suolo analizzati
ed è indicativo,
non solo della
concentrazione
assoluta dei
microrganismi
specifici che
si stanno
quantificando,
ma anche della
loro relativa
abbondanza
rispetto al
numero totale di batteri presenti
nel sito contaminato (Figura 2).
Nel caso specifico
dell’analisi dei microrganismi
decloranti, sono disponibili
sonde oligonucleotidiche
per l’identificazione e la
quantificazione mediante
Fish e Card-Fish sia i
principali decloranti parziali
(Desulfitobacterium,
Dehalobacter, Desulfuromonas,
Sulfurospirillum, Geobacter) che
il Dehalococcoides.
Il campionamento per l’analisi
Fish e Card-Fish è molto
semplice e prevede il prelievo
di acqua e/o suolo in quantità
variabili in base al campione
da analizzare (per la matrice
acquosa volume variabile da
min. 1 mL a max. 500 mL; per
il suolo, peso variabile da min.
1 g a max. 10 g). I tempi di
analisi sono rapidi e, in seguito
a una fase di fissaggio del
campione fresco in formaldeide
e filtrazione su appositi filtri
(max. 3 h), è possibile eseguire
l’ibridazione e ottenere il dato
quantitativo finale entro le 24 h
successive. Il campione filtrato
si può conservare per diversi
mesi e può essere utilizzato per
successive analisi.
Quantificazione mediante
qPCR e RT-qPCR
In aggiunta all’analisi
microscopica, che consente
una quantificazione puntuale
della concentrazione dei
microrganismi presenti nel
campione analizzato, è possibile
eseguire una stima quantitativa
di particolari specie microbiche
e dei rispettivi enzimi chiave di
interesse mediante la stima dei
geni (DNA) e del loro livello di
espressione (mRNA). Nel primo
caso, la tecnica elettiva è la
Real Time PCR (qPCR), mentre
nel secondo caso si adotta la
Reverse Transcription Real Time
PCR (RT-qPCR): entrambe sono
tecniche quantitative basate
sull’analisi di specifici geni di
biomarker di contaminazione (ad
esempio Dehalococcoides) e
della loro attività metabolica.
Nel dettaglio dei microrganismi
decloranti, la qPCR consente di
quantificare il Dehalococcoides
presente nei campioni analizzati,
mentre la RT-qPCR consente di
definire esattamente il livello di
attività declorante mediante la
stima quantitativa del livello di
espressione delle dealogenasi
riduttive, quest’ultimo da
considerarsi un parametro
molto importante per definire
il potenziale biodegradativo
di un sito contaminato. Il dato
quantitativo che si ottiene
dalla qPCR si esprime in
numero (n°) di copie di geni/
litro (L) acqua e/o grammo (g)
suolo mentre con l’impiego
della RT-qPCR si ottengono il
numero (n°) trascritti/litro (L)
acqua e/o grammo (g) suolo
oppure è possibile esprimere
il livello di attività metabolica
come numero (n°) trascritti/
numero (n°) copie gene. Il
campionamento per l’analisi
qPCR ed RT-qPCR prevede
una conservazione adeguata
dei campioni (-20 °C e -80 °C)
da cui è necessario eseguire
l’estrazione del DNA e dell’RNA
rispettivamente. È necessario
impiegare volumi maggiori
rispetto a quelli necessari
per la Fish e Card-Fish (da
1 a 5 litri di acqua di falda) e
filtrare i campioni già durante
la fase di campionamento per
preservare l’integrità del DNA e
dell’RNA. I tempi di analisi per
la quantificazione del numero
di copie di geni (qPCR) o di
trascritti (RT-qPCR) sono molto
rapidi ed è possibile ottenere il
dato quantitativo in meno di 24 h
dal campionamento.
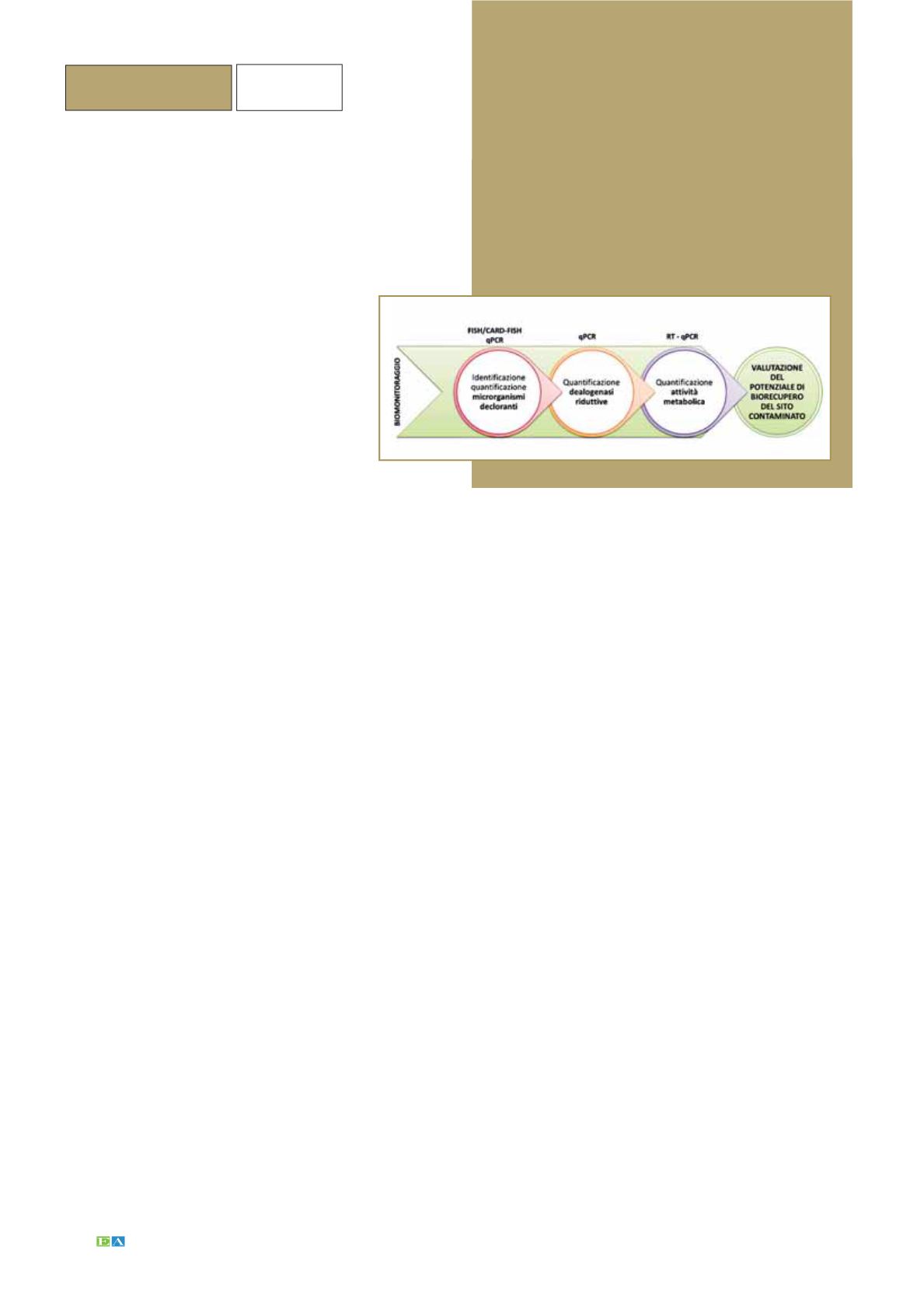
In seguito a studi di valutazione,
ottimizzazione e validazione
delle diverse tecniche
biomolecolari disponibili,
l’utilizzo combinato degli
approcci descritti si è dimostrato
essere il più vantaggioso
ai fini della valutazione
del biorecupero di un sito
contaminato da solventi clorurati
(Figura 3).
Uno dei vantaggi
BONIFICA
SUOLI
Figura 3 - Biomonitoraggio di siti contaminati da solventi
clorurati: tecniche biomolecolari applicate su campioni
ambientali (acqua di falda e/o suolo) per identi¿care,
quanti¿care e stimare il livello di attività metabolica di
microrganismi decloranti coinvolti nella degradazione
biologica di composti tossici


